- 1 Load packages
- 2 Forschungsfrage
- 3 Simulierte Daten
- 4 Differenzwert berechnen
- 5 Visualisieren
- 6 Deskriptive Statistik
- 7 Deskriptive Statistik als schöne Tabelle
- 8 Cohens d
- 9 Inferenzstatistik
- 10 Parameter (Koeffizienten des Modells) plotten
- 11 Ja, ist der Effekt jetzt groß oder nicht?
- 12 ROPE
- 13 Was ist mit R-Quadrat?
- 14 Fazit
- 15 Reproducibility
1 Load packages
library(tidyverse) # data wrangling
library(easystats) # make stasts easy again
library(rstanarm) # Bayes
library(gt) # schöne Tabellen2 Forschungsfrage
Stellen Sie sich vor, Sie haben ein Experiment durchgeführt. Im Zuge dessen haben Sie vor einer Intervention und nach einer Intervention die interessierende Variable (AV) gemessen. Jetzt sind sie an der Frage interessiert, ob die Veränderung in der AV sich zwischen den Experimentalbedingungen unterscheidet.
Ein Beispiel, um es konkreter zu machen. Sie sind interessiert am Effekt von Koffein auf die Konzentrationsfähigkeit. Dazu messen Sie die Konzentrationsfähigkeit (mit einem geeigneten Verfahren) vor und nach der experimentellen Intervention. Die experimentelle Intervention ist in diesem Fall die Gabe von 300mg Koffein in der Kontrollgruppe, und ein Placebo (kein Koffein) in der Kontrollgruppe.
Natürlich haben Sie auf möglichst gleiche Bedingungen zwischen den Gruppen und für alle Studienteilnehmer:innen geachtet. Außerdem haben Sie Störeinflüsse vermieden. Sogar die Stichprobengröße war ziemlich groß.
Okay. Was jetzt? Wie wertet man diese Daten aus (mit R)?
3 Simulierte Daten
Basteln wir uns einen Datensatz mit Fake-Daten:
d <-
tibble(
konz_t1 = rnorm(60),
konz_t2 = c(rnorm(30), rnorm(30, mean = 1)),
gruppe = c(rep("Placebo", 30), rep("Exp", 30)))Wir haben uns \(n=60\) Daten simuliert, 30 pro Gruppe (Experimental- und Placebogruppe). Zu \(t1\), vor dem Experiment, haben beide Populationen den gleichen Mittelwert, nämlich 0. Wir haben alle Werte aus einer Normalverteilung mit \(\mu=0, \sigma=1\) gezogen. Bei \(t2\) haben wir die Werte so simuliert, dass die Experimentalgruppe jetzt aus einer Normalverteilung mit \(\mu=1\) gezogen wird.
Das sieht dann so aus (die ersten paar Zeilen):
d %>%
head()
#> # A tibble: 6 × 3
#> konz_t1 konz_t2 gruppe
#> <dbl> <dbl> <chr>
#> 1 0.0315 0.507 Placebo
#> 2 0.540 -2.30 Placebo
#> 3 0.732 0.708 Placebo
#> 4 0.0861 0.368 Placebo
#> 5 1.23 -2.34 Placebo
#> 6 -2.54 0.418 PlaceboDas Simulieren der Daten ist bei einem echten Experiment natürlich nicht nötig. Durch das Messen bzw. das Durchführen des Experiments bekommen Sie die Daten “von allein”.
Beachten Sie, dass alle Daten in einer Tabelle stehen, nicht etwa in mehreren Tabellen.
4 Differenzwert berechnen
Als nächstes berechnen wir die Veränderung in der Konzentrationsfähigkeit für jede Person:
d2 <-
d %>%
mutate(differenz = konz_t2 - konz_t1)5 Visualisieren
Schauen wir uns da mal näher an:
d2 %>%
ggplot(aes(x = gruppe, y = differenz)) +
geom_violin(alpha = .5) +
geom_jitter(alpha = .7, width = .1) +
stat_summary(geom = "line", fun = mean, color = "red", group = 1)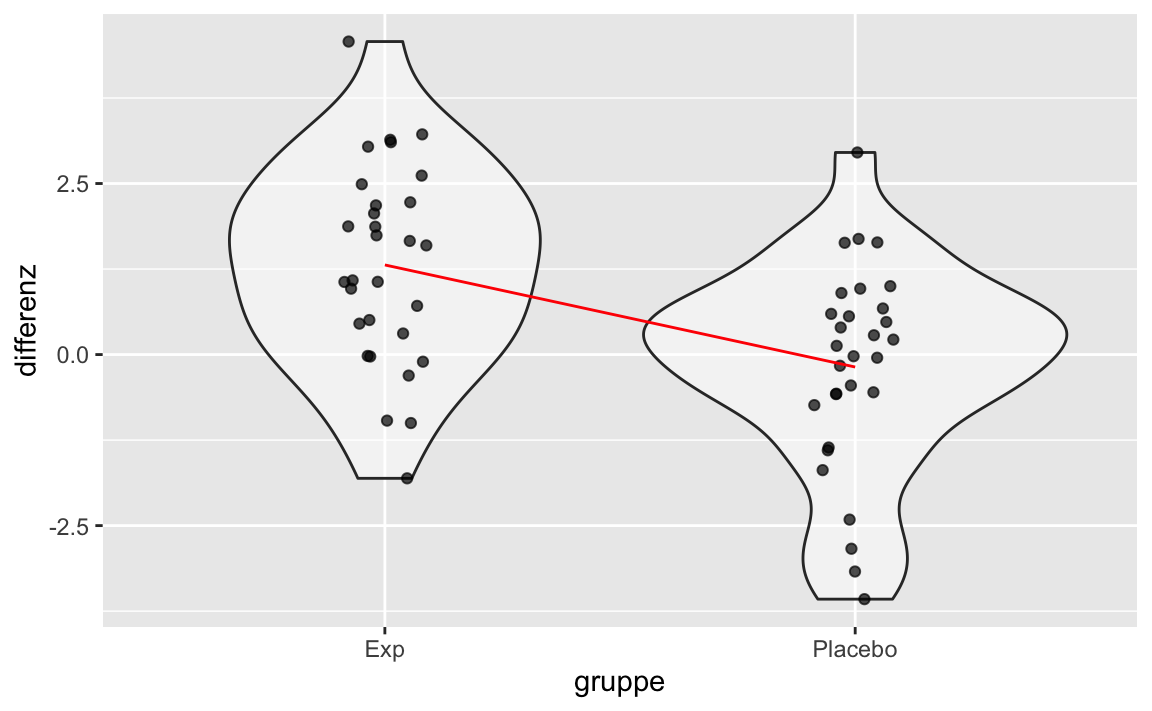
Wie man sieht, scheint der Zuwachs zwischen den Gruppen bei der Experimentalgruppe tatsächlich größer zu sein.
6 Deskriptive Statistik
d2 %>%
select(differenz, gruppe) %>%
group_by(gruppe) %>%
describe_distribution()
#> # gruppe=Exp
#>
#> Variable | Mean | SD | IQR | Range | Skewness | Kurtosis | n | n_Missing
#> -------------------------------------------------------------------------------------
#> differenz | 1.31 | 1.46 | 2.07 | [-1.81, 4.57] | -0.04 | -0.20 | 30 | 0
#>
#> # gruppe=Placebo
#>
#> Variable | Mean | SD | IQR | Range | Skewness | Kurtosis | n | n_Missing
#> --------------------------------------------------------------------------------------
#> differenz | -0.18 | 1.50 | 1.62 | [-3.57, 2.95] | -0.51 | 0.28 | 30 | 07 Deskriptive Statistik als schöne Tabelle
d2 %>%
select(differenz, gruppe) %>%
group_by(gruppe) %>%
describe_distribution() %>%
gt() %>%
fmt_number(columns = where(is.numeric))| Variable | Mean | SD | IQR | Min | Max | Skewness | Kurtosis | n | n_Missing | .group |
|---|---|---|---|---|---|---|---|---|---|---|
| differenz | 1.31 | 1.46 | 2.07 | −1.81 | 4.57 | −0.04 | −0.20 | 30.00 | 0.00 | gruppe=Exp |
| differenz | −0.18 | 1.50 | 1.62 | −3.57 | 2.95 | −0.51 | 0.28 | 30.00 | 0.00 | gruppe=Placebo |
Mehr zum Paket {gt} findet sich hier.
8 Cohens d
cohens_d(differenz ~ gruppe, data = d2)
#> Cohen's d | 95% CI
#> ------------------------
#> 1.01 | [0.47, 1.54]
#>
#> - Estimated using pooled SD.Sie könnten schreiben:
Der Unterschied im Mittelwert zwischen den beiden Experimentalbedingungen wird auf 0.95 geschätzt, 95% KI [0.41, 1.48].
Äh, war 0.95 ein großer Effekt, laut Cohen?
interpret_cohens_d(0.95)
#> [1] "large"
#> (Rules: cohen1988)Ja!
9 Inferenzstatistik
Aber ist der Unterschied zwischen den Gruppen wirklich “signifikant”?
Bemühen wir etwas Inferenzstatistik.
m1 <- stan_glm(differenz ~ gruppe, data = d2, refresh = 0)
m1
#> stan_glm
#> family: gaussian [identity]
#> formula: differenz ~ gruppe
#> observations: 60
#> predictors: 2
#> ------
#> Median MAD_SD
#> (Intercept) 1.3 0.3
#> gruppePlacebo -1.5 0.4
#>
#> Auxiliary parameter(s):
#> Median MAD_SD
#> sigma 1.5 0.1
#>
#> ------
#> * For help interpreting the printed output see ?print.stanreg
#> * For info on the priors used see ?prior_summary.stanregOk, der mediane Effekt zwischen den Gruppen wird auf ca. -1.5 geschätzt.
Aber ein Konfidenzintervall wäre schön:
posterior_interval(m1, prob = .95)
#> 2.5% 97.5%
#> (Intercept) 0.7657152 1.8679423
#> gruppePlacebo -2.2549596 -0.7436646
#> sigma 1.2476414 1.7896489Ah, jetzt sehen wir, dass das 95%-PI auf ca. -2.3 bis -0.7 geschätzt wird. Es gibt also einen Effekt zwischen den Gruppen: Die Null (der “Null-Effekt”) ist nicht im Schätzbereich plausibler Werte enthalten. Ob der Effekt groß ist, ist eine andere Frage - keine statistische.
So geht es noch ein bisschen einfacher, wenn auch die Informationen etwas reichhaltiger sind:
library(easystats)
parameters(m1)
#> Parameter | Median | 95% CI | pd | % in ROPE | Rhat | ESS | Prior
#> ----------------------------------------------------------------------------------------------------
#> (Intercept) | 1.32 | [ 0.77, 1.87] | 100% | 0% | 1.000 | 3338.00 | Normal (0.56 +- 4.12)
#> gruppePlacebo | -1.50 | [-2.25, -0.74] | 100% | 0% | 1.000 | 3841.00 | Normal (0.00 +- 8.18)Was ist pd:
Compute the Probability of Direction (pd, also known as the Maximum Probability of Effect - MPE). It varies between 50% and 100% (i.e., 0.5 and 1) and can be interpreted as the probability (expressed in percentage) that a parameter (described by its posterior distribution) is strictly positive or negative (whichever is the most probable). It is mathematically defined as the proportion of the posterior distribution that is of the median’s sign. Although differently expressed, this index is fairly similar (i.e., is strongly correlated) to the frequentist p-value.
pd gibt uns die Wahrscheinlichkeitsmasse links von der Null (da der Medianschätzwert negativ ist, sonst würde sich der Wert auf rechts von der Null beziehen).
Rhat und ESS sagen uns, ob das Stichprobenziehen gut funktioniert hat. Ja, sieht hier alles gut aus.
10 Parameter (Koeffizienten des Modells) plotten
Für parameters() gibt es auch eine plot-Methode:
plot(parameters(m1))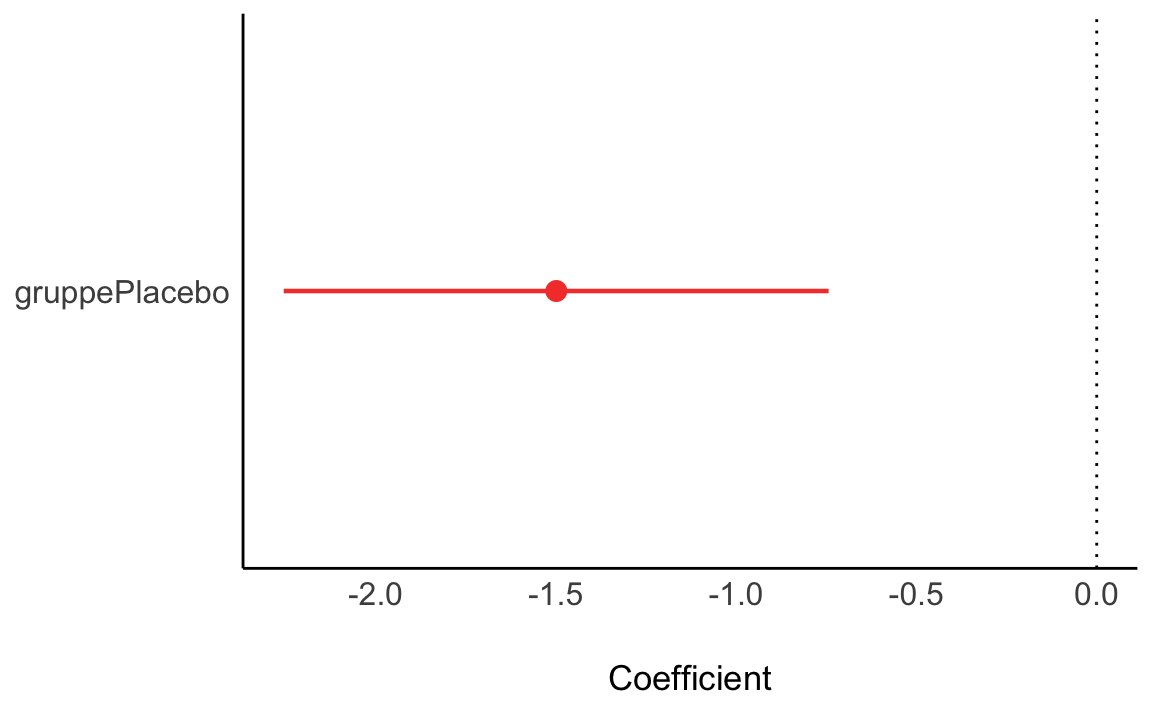
Alternativ bietet rstanarm auch eine Plot-Methoden:
plot(m1)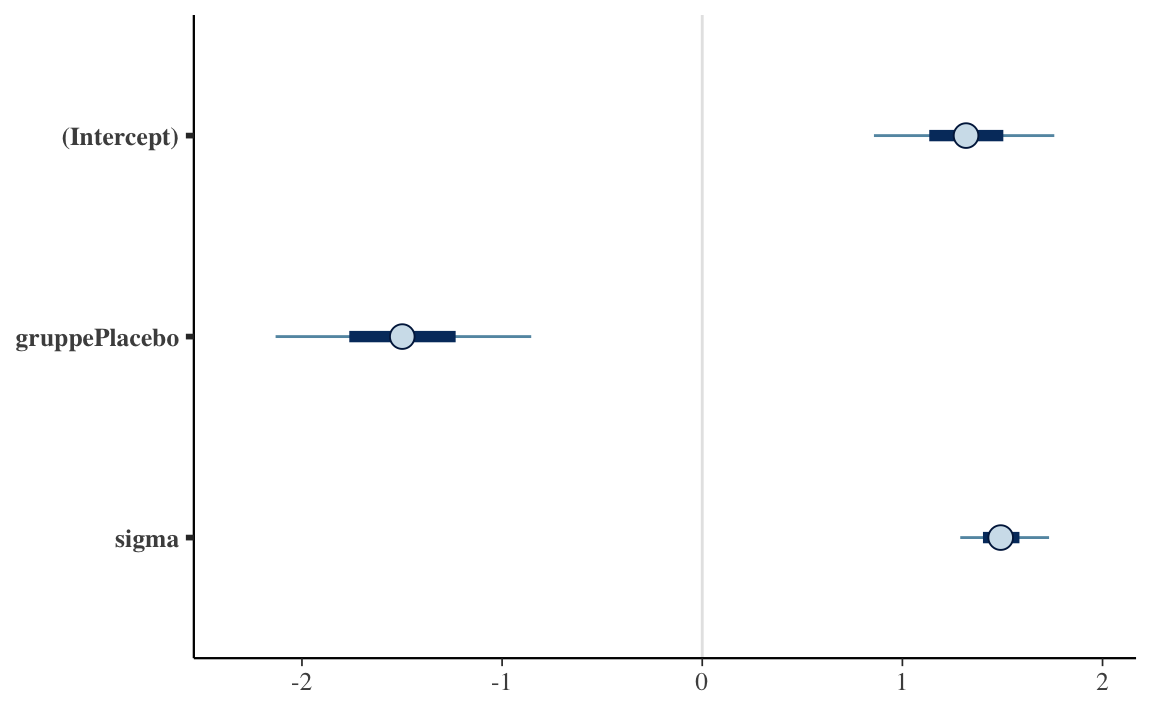
11 Ja, ist der Effekt jetzt groß oder nicht?
Also gut, definieren wir einen “gerade noch relevanten Effekt” auf 0.1 SD-Einheiten der AV (differenz).
Diese Wahl basiert auf Cohen (1988), vgl. Kruschke (2018).
z-Transformieren wir noch unsere AV, differenz,
dann können wir komfortabel schauen, ob der Effekt größer ist als ±0.1:
library(easystats)
d2 <-
d2 %>%
mutate(differenz_z = standardise(differenz))m2 <- stan_glm(differenz_z ~ gruppe, data = d2, refresh = 0)
coef(m2)
#> (Intercept) gruppePlacebo
#> 0.4540214 -0.9044253Ja, der Effekt ist deutlich größer als 0.1! Damit ist der Effekt größer als “vernachlässigbar klein”.
12 ROPE
Wir könnten noch ein ROPE berechnen, das sollte uns das gleiche wie oben bestätigen:
rope(m2)
#> # Proportion of samples inside the ROPE [-0.10, 0.10]:
#>
#> Parameter | inside ROPE
#> ---------------------------
#> (Intercept) | 0.00 %
#> gruppePlacebo | 0.00 %0% im Rope! Das Rope kann ausgeschlossen werden.
plot(rope(m2))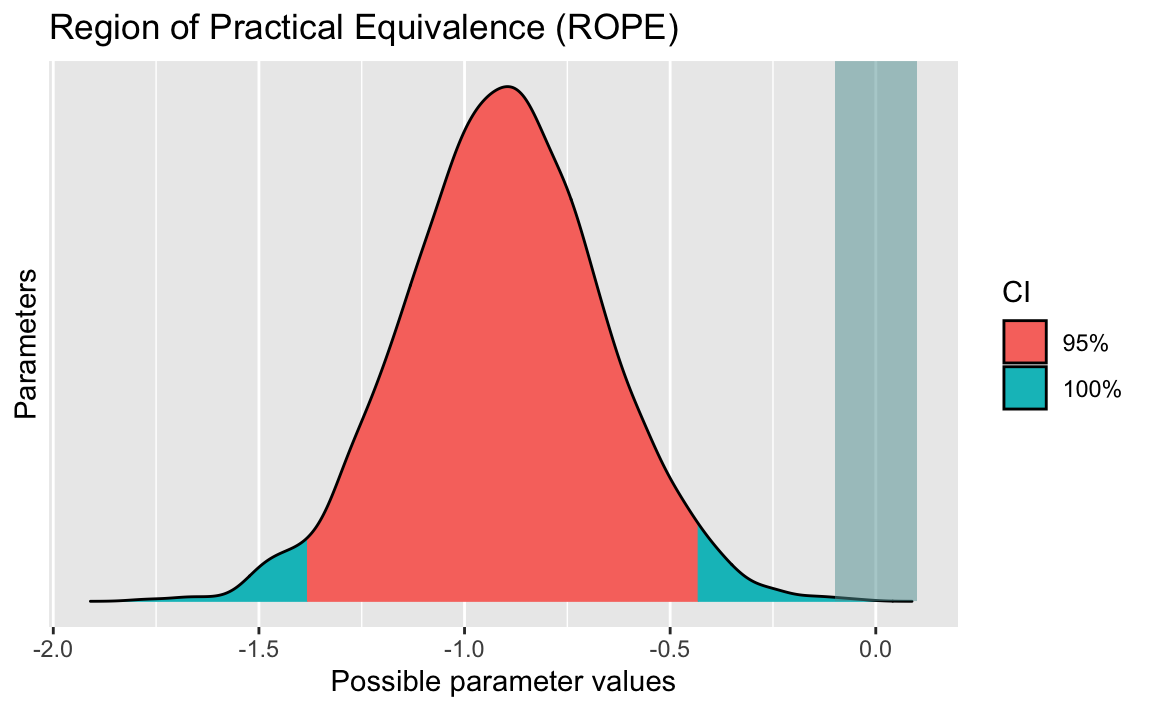
13 Was ist mit R-Quadrat?
Also gut, hier kommt noch \(R^2\):
r2_bayes(m2)
#> # Bayesian R2 with Compatibility Interval
#>
#> Conditional R2: 0.204 (95% CI [0.038, 0.364])14 Fazit
Wir resümieren, dass es offenbar einen Effekt gibt, und zwar einen, der größer ist als vernachlässigbar klein. Damit ist die “Nullhypothese”, die besagt, dass der Effekt Null ist, dass es also keinen Effekt gibt, ausgeschlossen. Kann man die Nullhypothese ausschließen, nennt man das Ergebnis auch “signifikant”. Zu beachten ist, dass der Begriff der Signifikanz verschiedene Bedeutungen haben kann. Hier definieren wir “signifikant”, wenn mind. 95% der Posteriori-Verteilung außerhalb des ROPE liegen (s. hier).
15 Reproducibility
#> ─ Session info ───────────────────────────────────────────────────────────────────────────────────────────────────────
#> setting value
#> version R version 4.1.3 (2022-03-10)
#> os macOS Big Sur/Monterey 10.16
#> system x86_64, darwin17.0
#> ui X11
#> language (EN)
#> collate en_US.UTF-8
#> ctype en_US.UTF-8
#> tz Europe/Berlin
#> date 2022-06-04
#>
#> ─ Packages ───────────────────────────────────────────────────────────────────────────────────────────────────────────
#> package * version date lib source
#> assertthat 0.2.1 2019-03-21 [1] CRAN (R 4.1.0)
#> backports 1.4.1 2021-12-13 [1] CRAN (R 4.1.0)
#> base64enc 0.1-3 2015-07-28 [1] CRAN (R 4.1.0)
#> bayesplot 1.8.1 2021-06-14 [1] CRAN (R 4.1.0)
#> bayestestR * 0.12.1 2022-05-02 [1] CRAN (R 4.1.3)
#> blogdown 1.8 2022-02-16 [2] CRAN (R 4.1.2)
#> bookdown 0.26.2 2022-05-02 [1] Github (rstudio/bookdown@6adacc3)
#> boot 1.3-28 2021-05-03 [2] CRAN (R 4.1.3)
#> brio 1.1.3 2021-11-30 [1] CRAN (R 4.1.0)
#> broom 0.8.0 2022-04-13 [1] CRAN (R 4.1.2)
#> bslib 0.3.1 2021-10-06 [1] CRAN (R 4.1.0)
#> cachem 1.0.6 2021-08-19 [1] CRAN (R 4.1.0)
#> callr 3.7.0 2021-04-20 [1] CRAN (R 4.1.0)
#> cellranger 1.1.0 2016-07-27 [1] CRAN (R 4.1.0)
#> cli 3.3.0 2022-04-25 [1] CRAN (R 4.1.2)
#> coda 0.19-4 2020-09-30 [1] CRAN (R 4.1.0)
#> codetools 0.2-18 2020-11-04 [2] CRAN (R 4.1.3)
#> colorout * 1.2-2 2022-01-04 [1] Github (jalvesaq/colorout@79931fd)
#> colorspace 2.0-3 2022-02-21 [1] CRAN (R 4.1.2)
#> colourpicker 1.1.0 2020-09-14 [2] CRAN (R 4.1.0)
#> correlation * 0.8.0.1 2022-04-21 [1] https://easystats.r-universe.dev (R 4.1.3)
#> crayon 1.5.1 2022-03-26 [1] CRAN (R 4.1.2)
#> crosstalk 1.1.1 2021-01-12 [2] CRAN (R 4.1.0)
#> datawizard * 0.4.0.17 2022-05-02 [1] Github (easystats/datawizard@de462e3)
#> DBI 1.1.2 2021-12-20 [1] CRAN (R 4.1.0)
#> dbplyr 2.1.1 2021-04-06 [1] CRAN (R 4.1.0)
#> desc 1.4.1 2022-03-06 [1] CRAN (R 4.1.2)
#> devtools 2.4.3 2021-11-30 [1] CRAN (R 4.1.0)
#> digest 0.6.29 2021-12-01 [1] CRAN (R 4.1.0)
#> dplyr * 1.0.9 2022-04-28 [1] CRAN (R 4.1.2)
#> DT 0.20 2021-11-15 [1] CRAN (R 4.1.0)
#> dygraphs 1.1.1.6 2018-07-11 [1] CRAN (R 4.1.0)
#> easystats * 0.4.3 2022-04-21 [1] https://easystats.r-universe.dev (R 4.1.3)
#> effectsize * 0.6.0.1 2022-01-26 [1] CRAN (R 4.1.2)
#> ellipsis 0.3.2 2021-04-29 [1] CRAN (R 4.1.0)
#> emmeans 1.7.3 2022-03-27 [1] CRAN (R 4.1.2)
#> estimability 1.3 2018-02-11 [1] CRAN (R 4.1.0)
#> evaluate 0.15 2022-02-18 [1] CRAN (R 4.1.2)
#> fansi 1.0.3 2022-03-24 [1] CRAN (R 4.1.2)
#> farver 2.1.0 2021-02-28 [1] CRAN (R 4.1.0)
#> fastmap 1.1.0 2021-01-25 [2] CRAN (R 4.1.0)
#> forcats * 0.5.1 2021-01-27 [1] CRAN (R 4.1.0)
#> fs 1.5.2 2021-12-08 [1] CRAN (R 4.1.0)
#> generics 0.1.2 2022-01-31 [1] CRAN (R 4.1.2)
#> ggplot2 * 3.3.6 2022-05-03 [1] CRAN (R 4.1.2)
#> ggridges 0.5.3 2021-01-08 [1] CRAN (R 4.1.0)
#> glue 1.6.2 2022-02-24 [1] CRAN (R 4.1.2)
#> gridExtra 2.3 2017-09-09 [2] CRAN (R 4.1.0)
#> gtable 0.3.0 2019-03-25 [1] CRAN (R 4.1.0)
#> gtools 3.9.2 2021-06-06 [2] CRAN (R 4.1.0)
#> haven 2.4.3 2021-08-04 [1] CRAN (R 4.1.0)
#> highr 0.9 2021-04-16 [1] CRAN (R 4.1.0)
#> hms 1.1.1 2021-09-26 [1] CRAN (R 4.1.0)
#> htmltools 0.5.2 2021-08-25 [1] CRAN (R 4.1.0)
#> htmlwidgets 1.5.4 2021-09-08 [1] CRAN (R 4.1.0)
#> httpuv 1.6.5 2022-01-05 [1] CRAN (R 4.1.2)
#> httr 1.4.3 2022-05-04 [1] CRAN (R 4.1.2)
#> igraph 1.2.11 2022-01-04 [1] CRAN (R 4.1.2)
#> inline 0.3.19 2021-05-31 [1] CRAN (R 4.1.0)
#> insight * 0.17.0.6 2022-05-02 [1] Github (easystats/insight@0e1dbaf)
#> jquerylib 0.1.4 2021-04-26 [1] CRAN (R 4.1.0)
#> jsonlite 1.8.0 2022-02-22 [1] CRAN (R 4.1.2)
#> knitr 1.39 2022-04-26 [1] CRAN (R 4.1.2)
#> labeling 0.4.2 2020-10-20 [1] CRAN (R 4.1.0)
#> later 1.3.0 2021-08-18 [1] CRAN (R 4.1.0)
#> lattice 0.20-45 2021-09-22 [2] CRAN (R 4.1.3)
#> lifecycle 1.0.1 2021-09-24 [1] CRAN (R 4.1.0)
#> lme4 1.1-29 2022-04-07 [1] CRAN (R 4.1.2)
#> loo 2.4.1 2020-12-09 [1] CRAN (R 4.1.0)
#> lubridate 1.8.0 2021-10-07 [1] CRAN (R 4.1.0)
#> magrittr 2.0.3 2022-03-30 [1] CRAN (R 4.1.2)
#> markdown 1.1 2019-08-07 [1] CRAN (R 4.1.0)
#> MASS 7.3-55 2022-01-16 [2] CRAN (R 4.1.3)
#> Matrix 1.4-0 2021-12-08 [2] CRAN (R 4.1.3)
#> matrixStats 0.61.0 2021-09-17 [1] CRAN (R 4.1.0)
#> memoise 2.0.0 2021-01-26 [2] CRAN (R 4.1.0)
#> mime 0.12 2021-09-28 [1] CRAN (R 4.1.0)
#> miniUI 0.1.1.1 2018-05-18 [1] CRAN (R 4.1.0)
#> minqa 1.2.4 2014-10-09 [1] CRAN (R 4.1.0)
#> modelbased * 0.8.0 2022-04-12 [1] https://easystats.r-universe.dev (R 4.1.3)
#> modelr 0.1.8 2020-05-19 [1] CRAN (R 4.1.0)
#> multcomp 1.4-19 2022-04-26 [2] CRAN (R 4.1.2)
#> munsell 0.5.0 2018-06-12 [1] CRAN (R 4.1.0)
#> mvtnorm 1.1-3 2021-10-08 [1] CRAN (R 4.1.0)
#> nlme 3.1-155 2022-01-16 [2] CRAN (R 4.1.3)
#> nloptr 2.0.0 2022-01-26 [1] CRAN (R 4.1.2)
#> parameters * 0.17.0.9 2022-04-20 [1] https://easystats.r-universe.dev (R 4.1.3)
#> performance * 0.9.0.2 2022-04-20 [1] https://easystats.r-universe.dev (R 4.1.3)
#> pillar 1.7.0 2022-02-01 [1] CRAN (R 4.1.2)
#> pkgbuild 1.2.0 2020-12-15 [2] CRAN (R 4.1.0)
#> pkgconfig 2.0.3 2019-09-22 [1] CRAN (R 4.1.0)
#> pkgload 1.2.4 2021-11-30 [1] CRAN (R 4.1.0)
#> plyr 1.8.7 2022-03-24 [1] CRAN (R 4.1.2)
#> prettyunits 1.1.1 2020-01-24 [1] CRAN (R 4.1.0)
#> processx 3.5.3 2022-03-25 [1] CRAN (R 4.1.2)
#> promises 1.2.0.1 2021-02-11 [2] CRAN (R 4.1.0)
#> ps 1.7.0 2022-04-23 [1] CRAN (R 4.1.2)
#> purrr * 0.3.4 2020-04-17 [1] CRAN (R 4.1.0)
#> R6 2.5.1 2021-08-19 [1] CRAN (R 4.1.0)
#> Rcpp * 1.0.8.3 2022-03-17 [1] CRAN (R 4.1.2)
#> RcppParallel 5.1.5 2022-01-05 [1] CRAN (R 4.1.2)
#> readr * 2.1.2 2022-01-30 [1] CRAN (R 4.1.2)
#> readxl 1.3.1 2019-03-13 [1] CRAN (R 4.1.0)
#> remotes 2.4.0 2021-06-02 [2] CRAN (R 4.1.0)
#> report * 0.5.1.1 2022-04-11 [1] https://easystats.r-universe.dev (R 4.1.3)
#> reprex 2.0.1 2021-08-05 [1] CRAN (R 4.1.0)
#> reshape2 1.4.4 2020-04-09 [1] CRAN (R 4.1.0)
#> rlang 1.0.2 2022-03-04 [1] CRAN (R 4.1.2)
#> rmarkdown 2.14 2022-04-25 [1] CRAN (R 4.1.2)
#> rprojroot 2.0.3 2022-04-02 [1] CRAN (R 4.1.2)
#> rsconnect 0.8.25 2021-11-19 [1] CRAN (R 4.1.0)
#> rstan 2.21.3 2021-12-19 [1] CRAN (R 4.1.0)
#> rstanarm * 2.21.1 2020-07-20 [1] CRAN (R 4.1.0)
#> rstantools 2.1.1 2020-07-06 [1] CRAN (R 4.1.0)
#> rstudioapi 0.13 2020-11-12 [1] CRAN (R 4.1.0)
#> rvest 1.0.2 2021-10-16 [1] CRAN (R 4.1.0)
#> sandwich 3.0-1 2021-05-18 [2] CRAN (R 4.1.0)
#> sass 0.4.1 2022-03-23 [1] CRAN (R 4.1.2)
#> scales 1.2.0 2022-04-13 [1] CRAN (R 4.1.3)
#> see * 0.7.0.1 2022-04-12 [1] https://easystats.r-universe.dev (R 4.1.3)
#> sessioninfo 1.1.1 2018-11-05 [2] CRAN (R 4.1.0)
#> shiny 1.7.1 2021-10-02 [1] CRAN (R 4.1.0)
#> shinyjs 2.0.0 2020-09-09 [2] CRAN (R 4.1.0)
#> shinystan 2.5.0 2018-05-01 [1] CRAN (R 4.1.0)
#> shinythemes 1.2.0 2021-01-25 [1] CRAN (R 4.1.0)
#> StanHeaders 2.21.0-7 2020-12-17 [1] CRAN (R 4.1.0)
#> stringi 1.7.6 2021-11-29 [1] CRAN (R 4.1.0)
#> stringr * 1.4.0 2019-02-10 [1] CRAN (R 4.1.0)
#> survival 3.2-13 2021-08-24 [2] CRAN (R 4.1.3)
#> testthat 3.1.4 2022-04-26 [1] CRAN (R 4.1.2)
#> TH.data 1.1-1 2022-04-26 [2] CRAN (R 4.1.2)
#> threejs 0.3.3 2020-01-21 [1] CRAN (R 4.1.0)
#> tibble * 3.1.7 2022-05-03 [1] CRAN (R 4.1.2)
#> tidyr * 1.2.0 2022-02-01 [1] CRAN (R 4.1.2)
#> tidyselect 1.1.2 2022-02-21 [1] CRAN (R 4.1.2)
#> tidyverse * 1.3.1 2021-04-15 [1] CRAN (R 4.1.0)
#> tzdb 0.1.2 2021-07-20 [2] CRAN (R 4.1.0)
#> usethis 2.0.1 2021-02-10 [2] CRAN (R 4.1.0)
#> utf8 1.2.2 2021-07-24 [1] CRAN (R 4.1.0)
#> vctrs 0.4.1 2022-04-13 [1] CRAN (R 4.1.2)
#> withr 2.5.0 2022-03-03 [1] CRAN (R 4.1.2)
#> xfun 0.30 2022-03-02 [1] CRAN (R 4.1.2)
#> xml2 1.3.3 2021-11-30 [1] CRAN (R 4.1.0)
#> xtable 1.8-4 2019-04-21 [1] CRAN (R 4.1.0)
#> xts 0.12.1 2020-09-09 [1] CRAN (R 4.1.0)
#> yaml 2.3.5 2022-02-21 [1] CRAN (R 4.1.2)
#> zoo 1.8-9 2021-03-09 [1] CRAN (R 4.1.0)
#>
#> [1] /Users/sebastiansaueruser/Library/R/x86_64/4.1/library
#> [2] /Library/Frameworks/R.framework/Versions/4.1/Resources/library