1 Setup
library(tidyverse)
library(mosaic)
library(testthat)
library(magrittr)
library(sessioninfo)
library(rstanarm)2 Fallbeispiel
Im Statistikskript der FOM-Hochschule (entwickelt vom ifes) wird folgendes Beispiel zur Einführung von simulationsbasierter Inferenz verwendet:
Im Rahmen einer Sonderveranstaltung der FOM Dortmund (6.10.2016) 102 und Münster (9.11.2017) tippten von n = 34 Teilnehmer*innen x = 12 im Rahmen eines Dreieckstest auf die richtige Probe, d. h. das andere Bier.
Wandeln wir die Daten leicht ab (dann geht es schöner auf).
Also sagen wir:
n <- 36
x <- 14
prob_null <- 1/3Aus Gründen der Konvergenz vergrößern wir die Stichprobe noch einmal:
n2 <- n * 10
x2 <- x * 103 Datensatz
Erstellen wir eine Funktion, die einen Datensatz, in Abhängigkeit gewählter Parameter und erstellt.
3.1 Funktion, um Daten zu simulieren
bier_data <- function(n, x) {
# n: sample size
# x: successes
# This function generates data for Bernoulli chain data
out <-
tibble(id = 1:n,
success = c(rep.int(1, x), rep.int(0, n-x)))
return(out)
}Probieren wir’s aus.
3.1.1 Kleine Stichprobe
d <- bier_data(n = n, x = x)
glimpse(d)
#> Rows: 36
#> Columns: 2
#> $ id <int> 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18,…
#> $ success <dbl> 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 0, 0, 0, 0, 0, 0, 0,…3.1.2 Große Stichprobe
d2 <- bier_data(n = n2, x = x2)3.2 Unit Testing
Im Sinne des Test-Driven-Designs: Schreiben wir zuerst einen Unit Test (oder mehrere) für die geplante (noch nicht geschriebene!) Funktion zur Erstellung der (simulierten) Daten.
test_that("Datensatz in Abhängigkeit von n und x erstellt",
{
expect_equivalent(nrow(bier_data(34, 12)), 34)
expect_equivalent(sum(bier_data(34,12)$success), 12)
})
#> Test passed 🥇4 Signifikanz-Tests
Wir testen also ungerichtet.
rührt daher, dass es drei Optionen gibt und nur eine davon ist ein Treffer.
4.1 Simulationsbasierte Inferenz (SBI)
4.1.1 Kleine Stichprobe
set.seed(1896)
Nullvtlg <- do(10000) * rflip(n = n, prob = prob_null)
glimpse(Nullvtlg)
#> Rows: 10,000
#> Columns: 4
#> $ n <dbl> 36, 36, 36, 36, 36, 36, 36, 36, 36, 36, 36, 36, 36, 36, 36, 36, …
#> $ heads <dbl> 10, 11, 9, 14, 10, 8, 13, 15, 8, 19, 11, 13, 10, 15, 15, 15, 14,…
#> $ tails <dbl> 26, 25, 27, 22, 26, 28, 23, 21, 28, 17, 25, 23, 26, 21, 21, 21, …
#> $ prop <dbl> 0.2777778, 0.3055556, 0.2500000, 0.3888889, 0.2777778, 0.2222222…Berechnen wir :
propdach <- sum(d$success) / nrow(d)
propdach
#> [1] 0.3888889Mit Methoden des tidyverse ist es umständlicher, propdach zu berechnen; das tidyverse ist für die Arbeit mit Tabellen ausgelegt, nicht für einzelne Werte (oder Vektoren).
Die Abweichung von prob_null in der Stichprobe beträgt:
abw_stipro <- abs(propdach - prob_null)
abw_stipro
#> [1] 0.05555556Diese Abweichung fügen wir bei jeder simulierten Stichprobe hinzu:
Nullvtlg <-
Nullvtlg %>% mutate(abw = abs(prop - prob_null))pvalue_simu1 <- prop( ~ (abw >= abw_stipro),
data = Nullvtlg)
pvalue_simu1
#> prop_TRUE
#> 0.48724.1.2 Große Stichprobe
Für die große Stichprobe analog:
set.seed(1896)
Nullvtlg2 <- do(10000) * rflip(n = n2, prob = prob_null)
Nullvtlg2 <-
Nullvtlg2 %>% mutate(abw = abs(prop - prob_null))
pvalue_simu2 <- prop( ~ (abw >= abw_stipro),
data = Nullvtlg2)
pvalue_simu2
#> prop_TRUE
#> 0.02614.2 -Test
Man beachte, dass der -Test nur zweiseitig testet.
4.2.1 Kleine Stichprobe
success_freq <- table(d$success)
success_freq
#>
#> 0 1
#> 22 14success_prob <- c(1 - prob_null, prob_null)pvalue_chi1 <- chisq.test(x = success_freq,
p = success_prob
)$p.value
pvalue_chi1
#> [1] 0.47950014.2.2 Große Stichprobe
pvalue_chi2 <-
chisq.test(x = table(d2$success),
p = success_prob)$p.value
pvalue_chi2
#> [1] 0.025347324.3 Binomialtest
4.3.1 kleine Stichprobe
pvalue_binom1 <- binom.test(x = x,
n = n,
p = prob_null)$p.value
pvalue_binom1
#> [1] 0.4829805Von Hand:
Der Erwartungswert einer binomialverteilten Zufallsvariablen ist so definiert:
, mit der Anzahl der Versuche und der Trefferwahrscheinlichkeit.
E_X <- n * 1/3
E_X
#> [1] 12Der Varianz einer binomialverteilten Zufallsvariablen ist so definiert:
, mit .
Var_X = n * 1/3 * 2/3
Var_X
#> [1] 8Bei genügend großen Stichproben ist normalverteilt, daher lässt sich ein z-Wert berechnen:
z <- (x - E_X) / sqrt(Var_X)
z
#> [1] 0.6666667Also beträgt der p-Wert:
1 - pnorm(q = z)
#> [1] 0.25249254.3.2 Große Stichprobe
pvalue_binom2 <- binom.test(x = x2,
n = n2,
p = prob_null)$p.value
pvalue_binom2
#> [1] 0.02908044.4 Logistische Regression
glm1 <- glm(success ~ 1,
data = d,
family = "binomial")
glm1 %>% summary()
#>
#> Call:
#> glm(formula = success ~ 1, family = "binomial", data = d)
#>
#> Deviance Residuals:
#> Min 1Q Median 3Q Max
#> -0.9925 -0.9925 -0.9925 1.3744 1.3744
#>
#> Coefficients:
#> Estimate Std. Error z value Pr(>|z|)
#> (Intercept) -0.4520 0.3419 -1.322 0.186
#>
#> (Dispersion parameter for binomial family taken to be 1)
#>
#> Null deviance: 48.114 on 35 degrees of freedom
#> Residual deviance: 48.114 on 35 degrees of freedom
#> AIC: 50.114
#>
#> Number of Fisher Scoring iterations: 4Zu beachten ist, dass Logitwerte zurückgegeben werden; diese müssen ggf. noch in Wahrscheinlichkeiten rückgerechnet werden.
exp(coef(glm1))
#> (Intercept)
#> 0.6363636logit2prob <- function(logit){
odds <- exp(logit)
prob <- odds / (1 + odds)
return(prob)
}logit2prob(coef(glm1))
#> (Intercept)
#> 0.3888889Etwas komfortabler vielleicht geht es mit predict:
predict(glm1, type = "response",
newdata = NULL)
#> 1 2 3 4 5 6 7 8
#> 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889
#> 9 10 11 12 13 14 15 16
#> 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889
#> 17 18 19 20 21 22 23 24
#> 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889
#> 25 26 27 28 29 30 31 32
#> 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889
#> 33 34 35 36
#> 0.3888889 0.3888889 0.3888889 0.3888889Allerdings bezieht sich der p-Wert hier auf eine Referenz-Wahrscheinlchkeit von , so dass der p-Wert nicht vergleichbar ist mit den obigen Analysen.
5 Bayes-Test mit gleichverteilter Priorverteilung und MCMC-Sampler
glm2 <- stan_glm(success ~ 1,
data = d,
prior = NULL,
refresh = 0,
family = "binomial")
glm2 %>% print()
#> stan_glm
#> family: binomial [logit]
#> formula: success ~ 1
#> observations: 36
#> predictors: 1
#> ------
#> Median MAD_SD
#> (Intercept) -0.4 0.4
#>
#> ------
#> * For help interpreting the printed output see ?print.stanreg
#> * For info on the priors used see ?prior_summary.stanregAuch hier bekommen wir einen Logit zurück, den wir ggf. in eine Wahrscheinlichkeit umrechnen wollen:
coef(glm2)
#> (Intercept)
#> -0.4339619logit2prob(coef(glm2))
#> (Intercept)
#> 0.39318075.1 Zwischen-Fazit
Da finden sich doch ein paar Abweichungen im p-Wert; die Tests testen nicht (ganz) das Gleiche. Möglich, dass die Voraussetzungen jeweils nicht (ausreichend) erfüllt sind. Bei simulationsbasierten Verfahren kommt natürlich noch die Stichproben-Varianz hinzu, die für Ungewissheit im Schätzer sorgt.
6 Konfidenz-Intervall
6.1 Simulationsbasierte Inferenz (SBI)
set.seed(1896)
Bootvtlg <- do(1e4) * prop( ~success,
data = resample(d),
success = 1)
glimpse(Bootvtlg)
#> Rows: 10,000
#> Columns: 1
#> $ prop_1 <dbl> 0.4166667, 0.3333333, 0.5277778, 0.3611111, 0.4444444, 0.388888…Die SD der Bootverteilung beträgt:
sd_boot <- sd(Bootvtlg$prop_1)
sd_boot
#> [1] 0.08153576ggplot(Bootvtlg) +
aes(x = prop_1) +
geom_bar()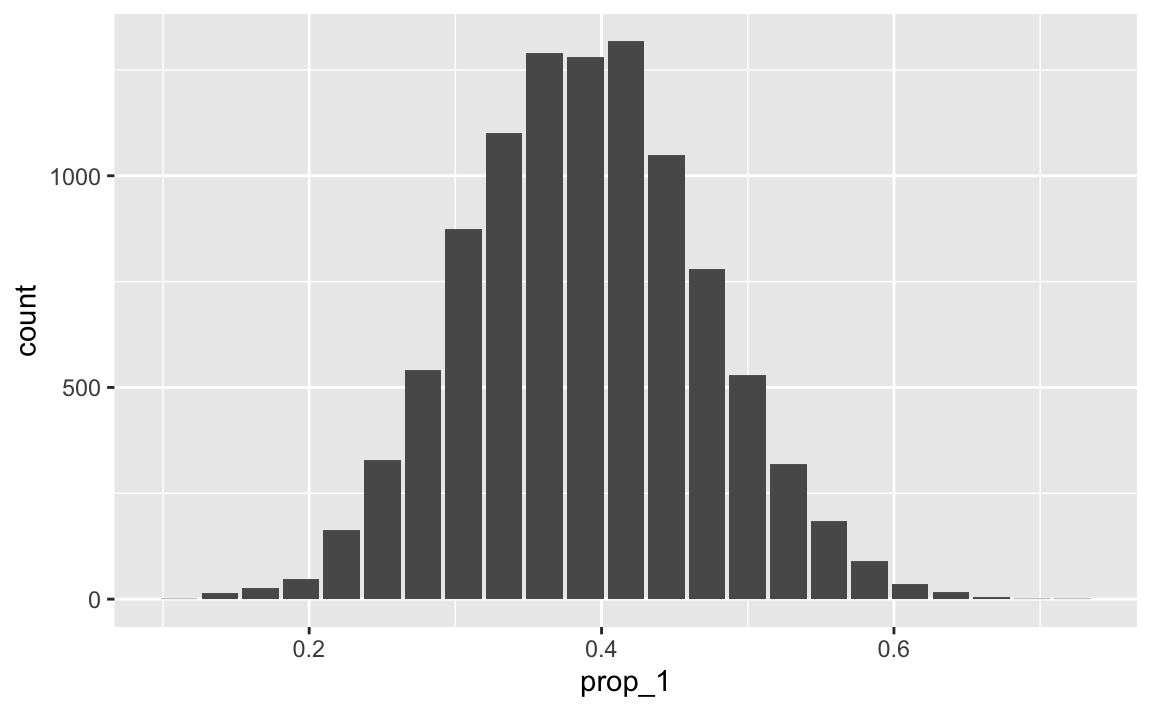
Wie man sieht, ist die Verteilung (annähernd) normalverteilt. D.h. man kann die Das Konfidenzintervall über die Näherung der Normalverteilungsquantile berechnen (s.u.).
KI-Grenzen:
ci95 <- quantile( ~ prop_1, data = Bootvtlg, probs = c(0.025, 0.975))
ci95
#> 2.5% 97.5%
#> 0.2222222 0.5555556Multipliziert man diese Anteilswerte mit , so bekommt man:
ci95[1]*n
#> 2.5%
#> 8
ci95[2]*n
#> 97.5%
#> 20Anders gesagt liegen die Intervallgrenzen bei 8 und 20.
6.2 -Test
Mit dem -Test kann man kein Konfidenzintervall für den Anteil bekommen, da die Prüfgröße nicht der Anteil ist.
6.2.1 Chi Square Bootstrap
Man könnte höchstens, wenn man unbedingt will, Die -Prüfgröße bootstrappen…
Also los:
chi_boot <- do(10000) * {chisq.test(x = table(resample(d$success)),
p = success_prob
) %>% extract2("statistic")}glimpse(chi_boot)
#> Rows: 10,000
#> Columns: 1
#> $ X.squared <dbl> 1.250000e-01, 1.250000e-01, 1.125000e+00, 5.000000e-01, 1.25…ggplot(chi_boot, aes(x = X.squared)) +
geom_histogram(bins = 20)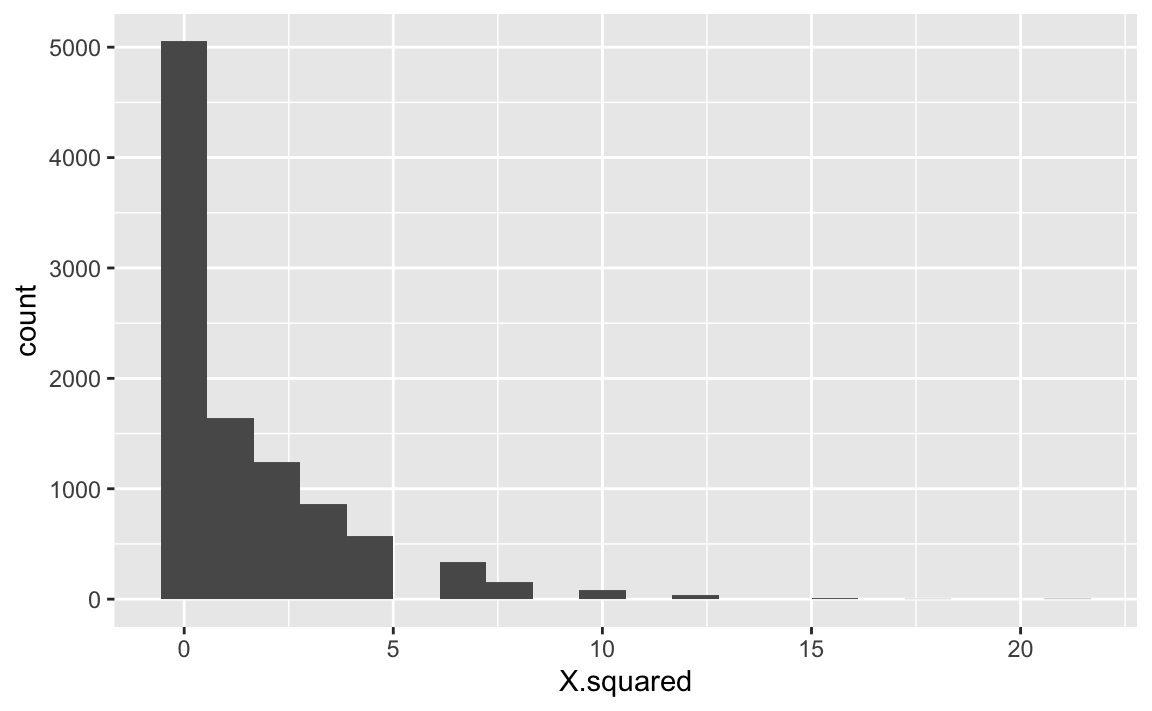
6.2.2 Chi p-value Bootstrap
In gleicher Manier kann man den p-Wert als Zufallsvariable auffassen und bootstrappen:
chi_p_boot <- do(10000) * {chisq.test(x = table(resample(d$success)),
p = success_prob
) %>% extract2("p.value")}glimpse(chi_p_boot)
#> Rows: 10,000
#> Columns: 1
#> $ result <dbl> 1.00000000, 0.15729921, 0.72367361, 0.47950012, 0.28884437, 0.4…ggplot(chi_p_boot, aes(x = result)) +
geom_histogram()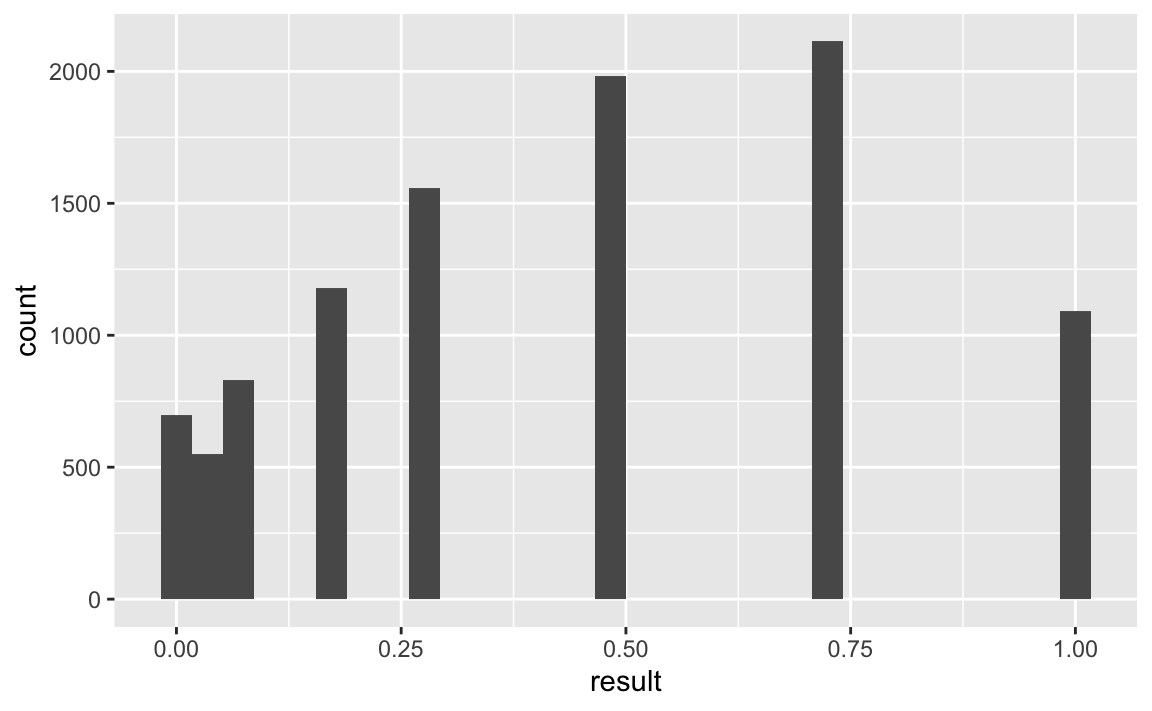
6.3 Binomial-Test, Näherung über Normalverteilung
Wie in der SBI gesehen, ist die Stichprobenverteilung bzw. die Bootstrapverteilung annähernd normalverteilt und zwar mit folgenden Parametern:
mue_boot <- x
sigma_boot <- sqrt(Var_X)Dieser theoretische Wert entspricht gut dem durch die Simulation gefundenen Wert:
sd_boot*n
#> [1] 2.9352886.4 Logistische Regression
Das kann man so machen (Quelle):
ci_glm1 <- predict(glm1, type = "response", se.fit = TRUE)
ci_glm1
#> $fit
#> 1 2 3 4 5 6 7 8
#> 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889
#> 9 10 11 12 13 14 15 16
#> 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889
#> 17 18 19 20 21 22 23 24
#> 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889
#> 25 26 27 28 29 30 31 32
#> 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889 0.3888889
#> 33 34 35 36
#> 0.3888889 0.3888889 0.3888889 0.3888889
#>
#> $se.fit
#> 1 2 3 4 5 6 7
#> 0.08124967 0.08124967 0.08124967 0.08124967 0.08124967 0.08124967 0.08124967
#> 8 9 10 11 12 13 14
#> 0.08124967 0.08124967 0.08124967 0.08124967 0.08124967 0.08124967 0.08124967
#> 15 16 17 18 19 20 21
#> 0.08124967 0.08124967 0.08124967 0.08124967 0.08124967 0.08124967 0.08124967
#> 22 23 24 25 26 27 28
#> 0.08124967 0.08124967 0.08124967 0.08124967 0.08124967 0.08124967 0.08124967
#> 29 30 31 32 33 34 35
#> 0.08124967 0.08124967 0.08124967 0.08124967 0.08124967 0.08124967 0.08124967
#> 36
#> 0.08124967
#>
#> $residual.scale
#> [1] 1Da wir keinen Prädiktor haben, wird für alle Fälle der gleiche Wert vorhergesagt.
Nun “bauen” wir das KI basierend auf der Annahme einer Normalverteilung:
critval <- 1.96 ## approx 95% CI
upr <- ci_glm1$fit + (critval * ci_glm1$se.fit)
lwr <- ci_glm1$fit - (critval * ci_glm1$se.fit)
lwr[1]
#> 1
#> 0.2296395
upr[1]
#> 1
#> 0.5481382Das sind die Anteilswerte; multiplizieren wir diese mit :
lwr[1]*n
#> 1
#> 8.267023
upr[1]*n
#> 1
#> 19.73298Also liegen die Intervallgrenzen etwa bei 8 (unten) bzw. 19.7329767 (oben).
6.5 Zwischen-Fazit
Die Werte der Konfidenzintervall-Grenzen sind ziemlich ähnlich; das überrascht nicht, denn die Bootverteilung ist schön normalverteilt; die Binomialmethode basiert ebenfalls auf einer Approximation der Normalverteilung. Daher resultieren hier ähnliche Werte.
7 Fazit
Die Bootstrap-Methode funktioniert hier gut; natürlich hat sie auch hier Fallstricke.
Der p-Wert unterscheidet sich je Methode, weil die Signifikanztests unterschiedliche Prüfgrößen berechnen, sprich verschiedene Dinge berechnen.
8 Reproducibility
#> ─ Session info ───────────────────────────────────────────────────────────────────────────────────────────────────────
#> setting value
#> version R version 4.1.0 (2021-05-18)
#> os macOS Big Sur 10.16
#> system x86_64, darwin17.0
#> ui X11
#> language (EN)
#> collate en_US.UTF-8
#> ctype en_US.UTF-8
#> tz Europe/Berlin
#> date 2021-07-29
#>
#> ─ Packages ───────────────────────────────────────────────────────────────────────────────────────────────────────────
#> package * version date lib source
#> assertthat 0.2.1 2019-03-21 [1] CRAN (R 4.1.0)
#> backports 1.2.1 2020-12-09 [1] CRAN (R 4.1.0)
#> blogdown 1.4 2021-07-23 [2] CRAN (R 4.1.0)
#> bookdown 0.22 2021-04-22 [2] CRAN (R 4.1.0)
#> broom 0.7.6 2021-04-05 [1] CRAN (R 4.1.0)
#> bslib 0.2.5.1 2021-05-18 [1] CRAN (R 4.1.0)
#> cellranger 1.1.0 2016-07-27 [1] CRAN (R 4.1.0)
#> cli 3.0.1 2021-07-17 [1] CRAN (R 4.1.0)
#> codetools 0.2-18 2020-11-04 [2] CRAN (R 4.1.0)
#> colorspace 2.0-2 2021-06-24 [1] CRAN (R 4.1.0)
#> crayon 1.4.1 2021-02-08 [1] CRAN (R 4.1.0)
#> crosstalk 1.1.1 2021-01-12 [2] CRAN (R 4.1.0)
#> DBI 1.1.1 2021-01-15 [1] CRAN (R 4.1.0)
#> dbplyr 2.1.1 2021-04-06 [1] CRAN (R 4.1.0)
#> digest 0.6.27 2020-10-24 [1] CRAN (R 4.1.0)
#> dplyr * 1.0.7 2021-06-18 [1] CRAN (R 4.1.0)
#> ellipsis 0.3.2 2021-04-29 [1] CRAN (R 4.1.0)
#> evaluate 0.14 2019-05-28 [1] CRAN (R 4.1.0)
#> fansi 0.5.0 2021-05-25 [1] CRAN (R 4.1.0)
#> farver 2.1.0 2021-02-28 [1] CRAN (R 4.1.0)
#> forcats * 0.5.1 2021-01-27 [1] CRAN (R 4.1.0)
#> fs 1.5.0 2020-07-31 [1] CRAN (R 4.1.0)
#> generics 0.1.0 2020-10-31 [1] CRAN (R 4.1.0)
#> ggdendro 0.1.22 2020-09-13 [1] CRAN (R 4.1.0)
#> ggforce 0.3.3 2021-03-05 [1] CRAN (R 4.1.0)
#> ggformula * 0.10.1 2021-01-13 [1] CRAN (R 4.1.0)
#> ggplot2 * 3.3.5 2021-06-25 [1] CRAN (R 4.1.0)
#> ggrepel 0.9.1 2021-01-15 [2] CRAN (R 4.1.0)
#> ggridges * 0.5.3 2021-01-08 [1] CRAN (R 4.1.0)
#> ggstance * 0.3.5 2020-12-17 [1] CRAN (R 4.1.0)
#> glue 1.4.2 2020-08-27 [1] CRAN (R 4.1.0)
#> gridExtra 2.3 2017-09-09 [2] CRAN (R 4.1.0)
#> gtable 0.3.0 2019-03-25 [1] CRAN (R 4.1.0)
#> haven 2.4.1 2021-04-23 [1] CRAN (R 4.1.0)
#> hms 1.1.0 2021-05-17 [1] CRAN (R 4.1.0)
#> htmltools 0.5.1.1 2021-01-22 [1] CRAN (R 4.1.0)
#> htmlwidgets 1.5.3 2020-12-10 [2] CRAN (R 4.1.0)
#> httr 1.4.2 2020-07-20 [1] CRAN (R 4.1.0)
#> jquerylib 0.1.4 2021-04-26 [1] CRAN (R 4.1.0)
#> jsonlite 1.7.2 2020-12-09 [1] CRAN (R 4.1.0)
#> knitr 1.33 2021-04-24 [1] CRAN (R 4.1.0)
#> labelled 2.8.0 2021-03-08 [2] CRAN (R 4.1.0)
#> lattice * 0.20-44 2021-05-02 [2] CRAN (R 4.1.0)
#> leaflet 2.0.4.1 2021-01-07 [1] CRAN (R 4.1.0)
#> lifecycle 1.0.0 2021-02-15 [1] CRAN (R 4.1.0)
#> lubridate 1.7.10 2021-02-26 [1] CRAN (R 4.1.0)
#> magrittr * 2.0.1 2020-11-17 [1] CRAN (R 4.1.0)
#> MASS 7.3-54 2021-05-03 [2] CRAN (R 4.1.0)
#> Matrix * 1.3-4 2021-06-01 [2] CRAN (R 4.1.0)
#> modelr 0.1.8 2020-05-19 [1] CRAN (R 4.1.0)
#> mosaic * 1.8.3 2021-01-18 [1] CRAN (R 4.1.0)
#> mosaicCore 0.9.0 2021-01-16 [1] CRAN (R 4.1.0)
#> mosaicData * 0.20.2 2021-01-16 [1] CRAN (R 4.1.0)
#> munsell 0.5.0 2018-06-12 [1] CRAN (R 4.1.0)
#> pillar 1.6.1 2021-05-16 [1] CRAN (R 4.1.0)
#> pkgconfig 2.0.3 2019-09-22 [1] CRAN (R 4.1.0)
#> plyr 1.8.6 2020-03-03 [1] CRAN (R 4.1.0)
#> polyclip 1.10-0 2019-03-14 [1] CRAN (R 4.1.0)
#> purrr * 0.3.4 2020-04-17 [1] CRAN (R 4.1.0)
#> R6 2.5.0 2020-10-28 [1] CRAN (R 4.1.0)
#> Rcpp 1.0.7 2021-07-07 [1] CRAN (R 4.1.0)
#> readr * 1.4.0 2020-10-05 [1] CRAN (R 4.1.0)
#> readxl 1.3.1 2019-03-13 [1] CRAN (R 4.1.0)
#> reprex 2.0.0 2021-04-02 [1] CRAN (R 4.1.0)
#> rlang 0.4.11 2021-04-30 [1] CRAN (R 4.1.0)
#> rmarkdown 2.9 2021-06-15 [1] CRAN (R 4.1.0)
#> rstudioapi 0.13 2020-11-12 [1] CRAN (R 4.1.0)
#> rvest 1.0.0 2021-03-09 [1] CRAN (R 4.1.0)
#> sass 0.4.0 2021-05-12 [1] CRAN (R 4.1.0)
#> scales * 1.1.1 2020-05-11 [1] CRAN (R 4.1.0)
#> sessioninfo * 1.1.1 2018-11-05 [2] CRAN (R 4.1.0)
#> stringi 1.7.3 2021-07-16 [1] CRAN (R 4.1.0)
#> stringr * 1.4.0 2019-02-10 [1] CRAN (R 4.1.0)
#> testthat * 3.0.4 2021-07-01 [2] CRAN (R 4.1.0)
#> tibble * 3.1.2 2021-05-16 [1] CRAN (R 4.1.0)
#> tidyr * 1.1.3 2021-03-03 [1] CRAN (R 4.1.0)
#> tidyselect 1.1.1 2021-04-30 [1] CRAN (R 4.1.0)
#> tidyverse * 1.3.1 2021-04-15 [1] CRAN (R 4.1.0)
#> tweenr 1.0.2 2021-03-23 [1] CRAN (R 4.1.0)
#> utf8 1.2.1 2021-03-12 [1] CRAN (R 4.1.0)
#> vctrs 0.3.8 2021-04-29 [1] CRAN (R 4.1.0)
#> withr 2.4.2 2021-04-18 [1] CRAN (R 4.1.0)
#> xfun 0.24 2021-06-15 [1] CRAN (R 4.1.0)
#> xml2 1.3.2 2020-04-23 [1] CRAN (R 4.1.0)
#> yaml 2.2.1.99 2021-06-18 [1] Github (viking/r-yaml@4788abe)
#>
#> [1] /Users/sebastiansaueruser/Library/R/x86_64/4.1/library
#> [2] /Library/Frameworks/R.framework/Versions/4.1/Resources/library