1 Setup
library(tidyverse)2 Daten laden
d <- read_csv("https://raw.githubusercontent.com/sebastiansauer/2021-sose/master/data/Impfbereitschaft/d3.csv")3 Von lang nach breit
d2 <-
d %>%
select(willingness:open2) %>%
pivot_longer(extra1:open2)d2 %>%
slice_head(n = 7)
#> # A tibble: 7 x 6
#> willingness health fear cases name value
#> <dbl> <dbl> <dbl> <dbl> <chr> <dbl>
#> 1 10 9 5 1 extra1 2
#> 2 10 9 5 1 agree1 2
#> 3 10 9 5 1 cons1 3
#> 4 10 9 5 1 neuro1 2
#> 5 10 9 5 1 open1 4
#> 6 10 9 5 1 extra2 1
#> 7 10 9 5 1 agree2 44 Plotten
d2 %>%
ggplot() +
aes(x = willingness, y = value) +
facet_wrap(~ name) +
geom_point() +
geom_smooth(method = "lm")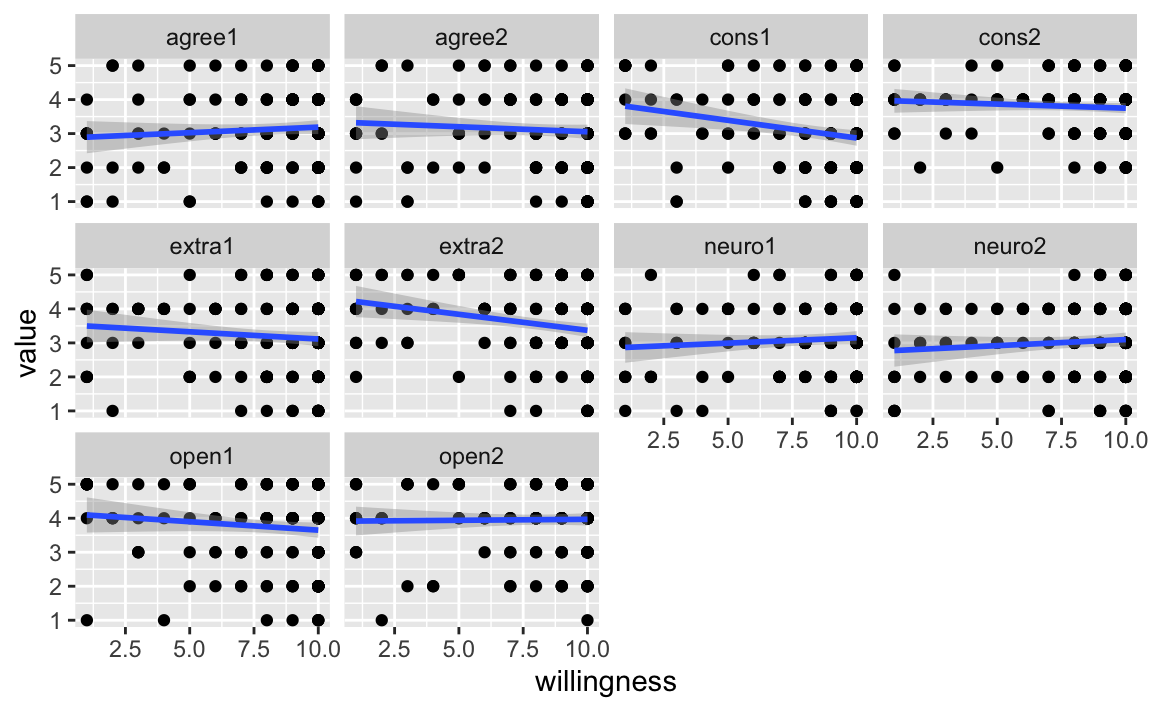
Jedes Diagramm zeigt den Zusammenhang von Impfbereitschaft mit einem Big-Five-Item. Das Diagramm ist alles andere als ideal; hier ging es nur darum, den Nutzen von pivot_longer() an einem praktischen Beispiel zu zeigen.
5 Kommentar
pivot_longer() ist nützlich, z.B. wenn man mehrere Variablen visualisieren möchte. Ein sinnvoller Zugang ist es dann, eine Facette pro Variable zu zeigen (s. Beispiel oben). Dazu erstellt man eine Gruppierungsvariable, die die verschiedenen Variablen als Werte fasst, z.B. die Werte “Variable1”, “Variable”, … . Dann kann man diese Gruppierungsvariable für die Facettierung nutzen. Wichtig: Man kann nur über eine Variable facettiereen (mit facet_wrap()). Daher muss man also die mehreren Variablen in eine umformatieren. Das erreicht man mit pivot_longer().