library(tidyverse)1 Was ist die Regression?
In diesem Post geht es um die einfache Regression (d.h. mit einem Prädiktor); genauer gesagt um die Frage, wie man auf die Formeln der Koeffizienten der einfachen Regression kommt.
Gehen wir von einigen zweidimensionalen Datenpunkten aus, die zu einem Phänomen gemessen wurden: \({(x_1, y_1), (x_2, y_2), \ldots, (x_n,y_)}\).
Gehen wir weiter davon aus, dass der Zusammenhang in Wirklichkeit von einer Geraden erklärt werden kann:
\[\hat{y}=b_0 + b_1x.\]
Eine Gerade kann durch zwei Koeffizienten definiert werden, den Achsenabschnitt \(b_0\) und die Steigung \(b_1\).
Für jeden Datenpunkt \(y_i\) können wir die Abweichung (den Fehler) zum vorhergesagten Wert \(\hat{y_i}\) ausrechnen:
\[e_i = y_i - \hat{y_i}\]
Nennen wir die Summe der quadrierten Fehler \(e_i\) im Folgenden \(QS\) (für Quadratsumme):
\[\begin{align*} QS &= \sum_{i=1}^{n}e_i^2\\ QS &= \sum_{i=1}^{n}(y_i - \hat{y_i})^2\\ QS &= \sum_{i=1}^{n}(y_i - b_0 - b_1x_i)^2 \end{align*}\]
Für eine übersichtlichere Notation wird der Index am Summenzeichen im Folgenden weggelassen.
Betrachten wir das an einem Datenbeispiel:
d <- mtcars
lm1 <- lm(mpg ~ hp, data = d)
d <-
d %>%
mutate(pred = predict(lm1),
resid = residuals(lm1))Jetzt visualisieren wir die Residuen (Abweichungen, Fehler):
ggplot(d,
aes(x = hp, y = mpg)) +
geom_smooth(method = "lm", se = FALSE, color = "dodgerblue") +
geom_segment(aes(xend = hp, yend = pred), alpha = .5) +
geom_point() +
geom_hline(yintercept = mean(d$mpg),
color = "grey60", linetype = "dashed") +
geom_vline(xintercept = mean(d$hp),
color = "grey60", linetype = "dashed") +
annotate("point", x = mean(d$hp), y = mean(d$mpg),
size = 5, color = "red", alpha = .5)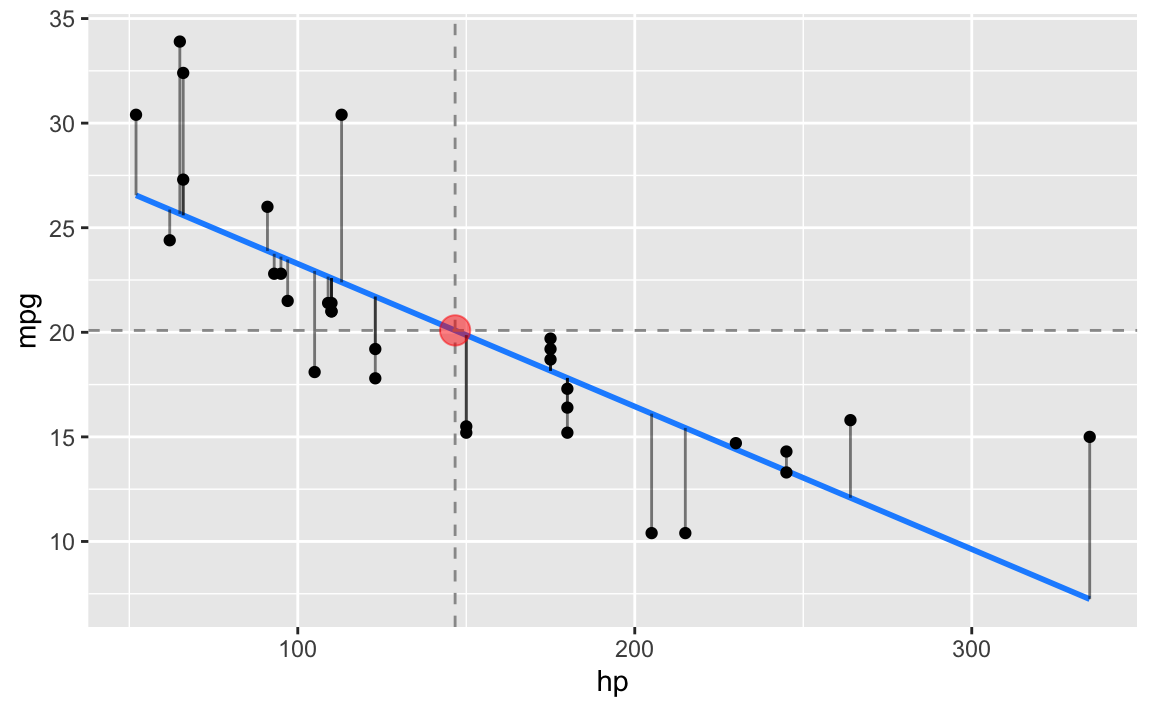
Die blaue Gerade ist die Regressionsgerade; die vertikalen Balken stellen die Residuen dar und die gestrichelten Linien repräsentieren jeweils die Mittelwerte von hp bzw. mpg. Man beachte, dass der Schnittpunkt der Mittelwertslinien auf der Regressionsgeraden liegt.
2 Wie findet man die Regressionsgerade?
Um die Koeffizienten der Regressionsgeraden zu bestimmen, können wir die Methode der kleinsten Quadrate (least squares) verwenden. Diese Methode gibt uns diejenigen Koeffizienten der Regressionsgeraden (also \(b_0\) und \(b_1\)), die die Quadratsumme (QS) der Residuen \(e_i\) minimieren.
Um die Quadratsumme zu minimieren, bilden wir jeweils die erste parzielle Ableitung und setzen diese Null. Anschließend löst man nach dem gesuchten Koeffizienten auf. Beginnen wir mit \(b_0\).
2.1 \(b_0\)
\[\begin{align*} \frac{\partial QS}{\partial b_0} &= 2 \sum(y_i - b_0 - b_1x_i)(-1) \\ &= -2 \sum y_i + 2 \sum b_0 + 2 \sum b_1 x_i \\ &= 2(-\sum y_i + nb_0 + b1 \sum x_i) = 0 \\ &= -\sum y_i + nb_0 + b1 \sum x_i = 0 \end{align*}\]
Der Term mit \(b_0\) bleibt auf der linken Seite, die anderen gehen auf die rechte Seite, damit wir nach dem gesuchten Koeffizienten auflösen können:
\[\begin{align*} n b_0&= \sum y_i - b_1 \sum x_i \\ b_0 &= \frac{\sum y_i - b_1 \sum x_i}{n} \\ &= n^{-1}\sum y_i - n^{-1}b_1 \sum x_i \\ &= \bar{y} - b_1 {x} \end{align*}\]
Das Ergebnis zeigt auch, dass der Punkt \((\bar{x}, \bar{y})\) auf der Regressionsgerade liegt.
2.2 \(b_1\)
Analog verfahren wir für den zweiten Koeffizienten, \(b_1\). Wieder bilden wir die parzielle Ableitung, setzen diese Null und lösen nach dem gesuchten Koeffizienten auf. Das Nachdifferenzieren (Kettenregel) liefert allerdings ein anderes Ergebnis.
\[\begin{align*} \frac{\partial QS}{\partial b_1} &= 2 \sum(y_i - b_0 - b_1x_i)(-x) \\ &=2(-\sum y_i x_i + b_0\sum x_i + b_1\sum x_i^2)\\ &=-2 \sum(x_iy_i - b_0x_i - b_1x_i^2) \end{align*}\]
Jetzt setzen wir \(b_0\) in die letzte Gleichung ein und setzen diese Null:
\[\begin{align*} &= -2 \sum(x_i y_i - (\bar{y} - b_1\bar{x})x_i - b_1x_i^2) \\ &= -2 \sum(x_iy_i - x_i\bar{y} + b_1 x_i \bar{x} - b_1x_i2) = 0 \end{align*}\]
die \(-2\) kürzt sich im Folgenden heraus. Um nach \(b_1\) aufzulösen, gruppieren wir die Terme in zwei Hälften: Die mit und die ohne \(b_1\):
\[ (x_i y_i - x_i\bar{y}) - b_1 \sum(x_i^2 - x_i \bar{x}) = 0 \]
Die Terme ohne \(b_1\) wandern auf die rechte Seite der Gleichung:
\[\begin{align*} b_1 \sum(x_i^2 - x_i \bar{x}) &= \sum(x_i y_i - x_i\bar{y}) \\ b_1 &= \frac{\sum x_i y_i - x_i \bar{y}}{\sum x_i^2 - x_i \bar{x}} = \\ &= \frac{x_i y_i - n\bar{x} \bar{y}}{\sum x_i^2 - n \bar{x}\bar{x}} \end{align*}\]
2.3 Weitere Umformung von \(b_1\)
Damit haben wir nach \(b_1\) aufgelöst. Allerdings kann man diesen letzten Ausdruck noch in bekanntere Terme umformulieren. Dazu sind zwei Hilfsüberlegungen nützlich:
2.3.1 Hilfsterm 1
\[ \sum (\bar{x}^2 - x_i \bar{x}) = n\bar{x}^2 - n \bar{x} \bar{x} = 0 \]
2.3.2 Hilfsterm 2
\[ \sum (\bar{x}\bar{y} - y_i \bar{x}) = n \bar{x} \bar{y} - n \bar{x} \bar{y} = 0 \]
Setzen wir diese Hilfsterme in obige Gleichung ein und zwar Hilfsterm 1 in den Nenner und Hilfsterm 2 in den Zähler. Das “dürfen” wir, da die Hilfsterme ja jeweils Null sind.
\[\begin{align*} b_1 &= \frac{(\sum x_i y_i - x_i \bar{y}) + \sum (\bar{x}\bar{y} - y_i \bar{x})}{(\sum x_i^2 - x_i \bar{x}) + \sum (\bar{x}^2 - x_i \bar{x})} \end{align*}\]
2.3.3 Umformung des Zählers
Im Folgenden betrachten wir der Einfachheit halber nur den Zähler.
\[\begin{align*} b_1 &= \sum x_i y_i - \sum x_i \bar{y} + \sum \bar{x} \bar{y} - \sum y_i \bar{x} \\ &= \sum(x_i y_i - x_i \bar{y} + \bar{x}\bar{y} - y_i \bar{x}) \\ &= \sum(x_i(y_i - \bar{y}) + \bar{x}(y_i - \bar{y})) \\ &= \sum \left( (x_i - \bar{x})(y_i - \bar{y}) \right) \end{align*}\]
Multipliziert man nun Zähler und Nenner jeweils (und damit neutral) mit \(1/n\), so erhält man
\[ b1 = \frac{cov(x,y)}{s^2(x)}. \]
Dabei steht \(s^2(x)\) für die Varianz von \(x\) und \(cov\) für die Kovarianz.
2.4 Noch weitere Umformung von \(b_1\)
\[\begin{align*} b1 &= \frac{cov(x,y)}{s^2(x)} \\ &= \frac{cov(x,y)}{s^2(x)} \cdot \frac{s(y)}{s(y)} \\ &= \frac{cov(x,y)}{s(x)s(y)} \cdot \frac{s(y)}{s(x)} \\ &= cor(x,y) \cdot \frac{s(y)}{s(x)} \end{align*}\]
3 Fazit
In diesem Post ging es um die Ableitung der Formeln der Regressionskoeffizienten, zumindest der einfachen Regression. Dabei wurden grundlagende Algebra und Parzielle Ableitungen verwendet. Wir haben also bewiesen, dass die Regressionskoeffizienten die Form haben, die sie haben.
Für die Ableitung der multiplen Regression sind Ansätze auf Basis der linearen Algebra praktischer.
4 Reproduzierbarkeit
#> ─ Session info ───────────────────────────────────────────────────────────────────────────────────────────────────────
#> setting value
#> version R version 4.0.2 (2020-06-22)
#> os macOS 10.16
#> system x86_64, darwin17.0
#> ui X11
#> language (EN)
#> collate en_US.UTF-8
#> ctype en_US.UTF-8
#> tz Europe/Berlin
#> date 2021-04-07
#>
#> ─ Packages ───────────────────────────────────────────────────────────────────────────────────────────────────────────
#> package * version date lib source
#> assertthat 0.2.1 2019-03-21 [1] CRAN (R 4.0.0)
#> backports 1.2.1 2020-12-09 [1] CRAN (R 4.0.2)
#> blogdown 1.1 2021-01-19 [1] CRAN (R 4.0.2)
#> bookdown 0.21.6 2021-02-02 [1] Github (rstudio/bookdown@6c7346a)
#> broom 0.7.5 2021-02-19 [1] CRAN (R 4.0.2)
#> bslib 0.2.4.9000 2021-02-02 [1] Github (rstudio/bslib@b3cd7a9)
#> cachem 1.0.4 2021-02-13 [1] CRAN (R 4.0.2)
#> callr 3.5.1 2020-10-13 [1] CRAN (R 4.0.2)
#> cellranger 1.1.0 2016-07-27 [1] CRAN (R 4.0.0)
#> cli 2.3.1 2021-02-23 [1] CRAN (R 4.0.2)
#> codetools 0.2-16 2018-12-24 [2] CRAN (R 4.0.2)
#> colorspace 2.0-0 2020-11-11 [1] CRAN (R 4.0.2)
#> crayon 1.4.1 2021-02-08 [1] CRAN (R 4.0.2)
#> DBI 1.1.1 2021-01-15 [1] CRAN (R 4.0.2)
#> dbplyr 2.1.0 2021-02-03 [1] CRAN (R 4.0.2)
#> debugme 1.1.0 2017-10-22 [1] CRAN (R 4.0.0)
#> desc 1.2.0 2018-05-01 [1] CRAN (R 4.0.0)
#> devtools 2.3.2 2020-09-18 [1] CRAN (R 4.0.2)
#> digest 0.6.27 2020-10-24 [1] CRAN (R 4.0.2)
#> dplyr * 1.0.5 2021-03-05 [1] CRAN (R 4.0.2)
#> ellipsis 0.3.1 2020-05-15 [1] CRAN (R 4.0.0)
#> evaluate 0.14 2019-05-28 [1] CRAN (R 4.0.0)
#> fansi 0.4.2 2021-01-15 [1] CRAN (R 4.0.2)
#> fastmap 1.1.0 2021-01-25 [1] CRAN (R 4.0.2)
#> forcats * 0.5.1 2021-01-27 [1] CRAN (R 4.0.2)
#> fs 1.5.0 2020-07-31 [1] CRAN (R 4.0.2)
#> generics 0.1.0 2020-10-31 [1] CRAN (R 4.0.2)
#> ggplot2 * 3.3.3 2020-12-30 [1] CRAN (R 4.0.2)
#> glue 1.4.2 2020-08-27 [1] CRAN (R 4.0.2)
#> gtable 0.3.0 2019-03-25 [1] CRAN (R 4.0.0)
#> haven 2.3.1 2020-06-01 [1] CRAN (R 4.0.0)
#> hms 1.0.0 2021-01-13 [1] CRAN (R 4.0.2)
#> htmltools 0.5.1.1 2021-01-22 [1] CRAN (R 4.0.2)
#> httr 1.4.2 2020-07-20 [1] CRAN (R 4.0.2)
#> jquerylib 0.1.3 2020-12-17 [1] CRAN (R 4.0.2)
#> jsonlite 1.7.2 2020-12-09 [1] CRAN (R 4.0.2)
#> knitr 1.31 2021-01-27 [1] CRAN (R 4.0.2)
#> lifecycle 1.0.0 2021-02-15 [1] CRAN (R 4.0.2)
#> lubridate 1.7.9.2 2020-11-13 [1] CRAN (R 4.0.2)
#> magrittr 2.0.1 2020-11-17 [1] CRAN (R 4.0.2)
#> memoise 2.0.0 2021-01-26 [1] CRAN (R 4.0.2)
#> modelr 0.1.8 2020-05-19 [1] CRAN (R 4.0.0)
#> munsell 0.5.0 2018-06-12 [1] CRAN (R 4.0.0)
#> pillar 1.5.1 2021-03-05 [1] CRAN (R 4.0.2)
#> pkgbuild 1.2.0 2020-12-15 [1] CRAN (R 4.0.2)
#> pkgconfig 2.0.3 2019-09-22 [1] CRAN (R 4.0.0)
#> pkgload 1.2.0 2021-02-23 [1] CRAN (R 4.0.2)
#> prettyunits 1.1.1 2020-01-24 [1] CRAN (R 4.0.0)
#> processx 3.4.5 2020-11-30 [1] CRAN (R 4.0.2)
#> ps 1.5.0 2020-12-05 [1] CRAN (R 4.0.2)
#> purrr * 0.3.4 2020-04-17 [1] CRAN (R 4.0.0)
#> R6 2.5.0 2020-10-28 [1] CRAN (R 4.0.2)
#> Rcpp 1.0.6 2021-01-15 [1] CRAN (R 4.0.2)
#> readr * 1.4.0 2020-10-05 [1] CRAN (R 4.0.2)
#> readxl 1.3.1 2019-03-13 [1] CRAN (R 4.0.0)
#> remotes 2.2.0 2020-07-21 [1] CRAN (R 4.0.2)
#> reprex 1.0.0 2021-01-27 [1] CRAN (R 4.0.2)
#> rlang 0.4.10 2020-12-30 [1] CRAN (R 4.0.2)
#> rmarkdown 2.7 2021-02-19 [1] CRAN (R 4.0.2)
#> rprojroot 2.0.2 2020-11-15 [1] CRAN (R 4.0.2)
#> rstudioapi 0.13.0-9000 2021-02-11 [1] Github (rstudio/rstudioapi@9d21f50)
#> rvest 0.3.6 2020-07-25 [1] CRAN (R 4.0.2)
#> sass 0.3.1 2021-01-24 [1] CRAN (R 4.0.2)
#> scales 1.1.1 2020-05-11 [1] CRAN (R 4.0.0)
#> sessioninfo 1.1.1 2018-11-05 [1] CRAN (R 4.0.0)
#> stringi 1.5.3 2020-09-09 [1] CRAN (R 4.0.2)
#> stringr * 1.4.0 2019-02-10 [1] CRAN (R 4.0.0)
#> testthat 3.0.2 2021-02-14 [1] CRAN (R 4.0.2)
#> tibble * 3.1.0 2021-02-25 [1] CRAN (R 4.0.2)
#> tidyr * 1.1.3 2021-03-03 [1] CRAN (R 4.0.2)
#> tidyselect 1.1.0 2020-05-11 [1] CRAN (R 4.0.0)
#> tidyverse * 1.3.0 2019-11-21 [1] CRAN (R 4.0.0)
#> usethis 2.0.1 2021-02-10 [1] CRAN (R 4.0.2)
#> utf8 1.1.4 2018-05-24 [1] CRAN (R 4.0.0)
#> vctrs 0.3.6 2020-12-17 [1] CRAN (R 4.0.2)
#> withr 2.4.1 2021-01-26 [1] CRAN (R 4.0.2)
#> xfun 0.22 2021-03-11 [1] CRAN (R 4.0.2)
#> xml2 1.3.2 2020-04-23 [1] CRAN (R 4.0.0)
#> yaml 2.2.1 2020-02-01 [1] CRAN (R 4.0.0)
#>
#> [1] /Users/sebastiansaueruser/Rlibs
#> [2] /Library/Frameworks/R.framework/Versions/4.0/Resources/library